Faktabank
Hur får man fram inavelskoefficienter?
Hästen har 64 kromosomer totalt, varav 31 kromosompar samt könskromosomerna (XX eler XY). Kromosomerna förekommer parvis eftersom varje individ ärver den ena från sin far och den andra från sin mor. Det innebär också att det finns två kopior av de flesta anlag. Om dessa är lika säger man att de är homozygota och är de olika kallas de heterozygota.

Inavelskoefficienterna, som ofta presenteras i %, beräknas på individnivå som sannolikheten att två alleler (varianter ärvda från mor respektive far) på någon plats (”locus”) i DNA hos en individ är identiska genom arv. De kan bara var identiska genom arv om de kommer från en gemensam ana (förfader) till en individs mor och far. Vi talar här om sannolikheter eftersom det vid bildningen av könsceller (spermier och ägg) finns en slumpeffekt för vilken variant av de två anlag som varje förälder bär på, som nedärvs till avkomman.
I exemplet nedan så ärvde individen X två alleler som var lika genom arv (från A via föräldrarna B och C), men X hade också kunnat ärva två olika varianter.

Eftersom det finns så väldigt många ställen i genomet där detta kan ske (hästen har mer än 20 tusen kända gener och därtill finns ändå mer DNA-kod som inte kodar för proteiner) så fungerar sannolikhetsberäkningarna bra när man utgår ifrån släktskap baserat på härstamningsinformation. Det förutsätter förstås att informationen är korrekt och komplett. Att beräkna inavelsgrad utifrån härstamningsinformation har gjorts under lång tid för många olika djurpopulationer. Om man jämför medel-inavelsnivåer mellan olika populationer måste man dock se till hur djup härstamningsinformation (antal generationer bakåt) som använts, eftersom det påverkar nivån.
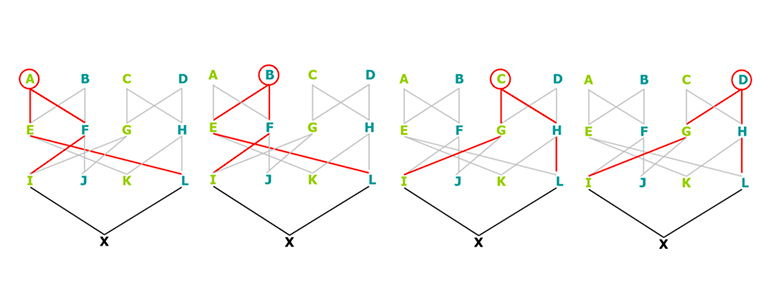
Sannolikheten att en individ ärver båda allelerna i ett genpar från samma förfader ökar om den förfadern var en nära släkting till både far och mor, jämfört med om den finns längre bak i stamtavlan då släktskapet halveras för varje generation. Sannolikheten är också högre ju fler gånger den förfadern förekommer i stamtavlan hos både mor och far till en individ. Det kan förstås också vara flera olika gemensamma förfäder till far och mor som bidrar till en individs inavelskoefficient. Nedan är ett extremt exempel (för att spara plats och förenkla) på nära inavel på flera förfäder i stamtavlan.

Ju fler sådana ”loopar” i stamtavlan som leder till en gemensam förfader till både mor och far, desto högre inavelskoefficient, och är dessutom både mor och/eller far själva inavlade på dessa gemensamma förfäder så ökar inavelskoefficienten ytterligare.
I enkla fall kan man räkna själv genom att i stamtavlan gå från ena föräldern till den andra (via gemensam ana) och räkna alla individer (inklusive föräldrarna), och det antalet blir då n i formeln för inavelsgrad.
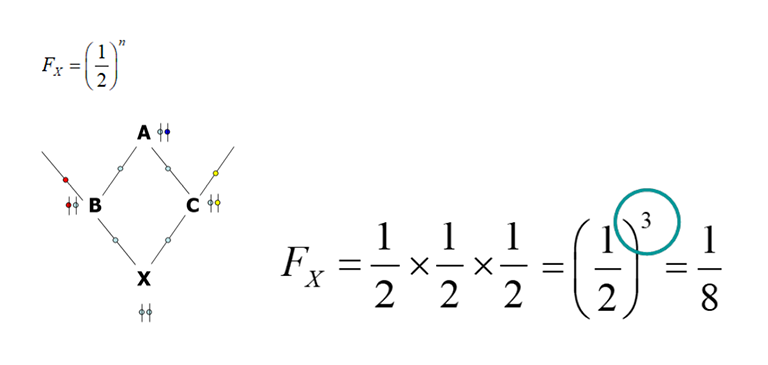
I det första exemplet blir då n=3 (förälder B, gemensam ana A, och förälder C) vilket motsvarar en inavelskoefficient på 12,5% hos individ X.
Det finns några olika sådana här räkneregler. Inavelsgraden hos en individ är detsamma som halva släktskapet mellan dess föräldrar, vilket innebär att man också kan räkna ut släktskapsgraden och utgå ifrån den. I exemplet ovan är t.ex. B och C halvsyskon med släktskapsgraden 0,25 och inavelsgraden hos deras gemensamma avkomma blir då hälften av det: 0,125, eller uttryckt som inavelskoefficient i procent 12,5%.
I Breedly räknas numer inavelskoefficienterna baserat på 7 generationer bakåt i härstamningen för kallblodstravare. När man som i detta fall tar med flera generationer bakåt och det finns många förfäder som förekommer flera gånger blir det komplicerat att räkna för hand, och för att effektivt skatta inavelskoefficienter för många individer på en gång (utan räknefel) behöver man kunna programmera detta. Det finns några olika metoder som använts och som generellt ger väldigt lika resultat, men som är olika snabba och ger olika flexibilitet.
Den metod som idag används i Breedly bygger på VanRadens ”tabular method” från 1992. Den är en lite långsammare metod men ger möjlighet att bestämma hur många generationer bakåt man vill räkna (i dagsläget 7). VanRadens metod har använts i flera olika kända analysprogram för att skatta inavelskoefficenter hos djur. Med denna metod (”tabular method”) utgår man ifrån en härstamningsfil med tre kolumner: individ, far och mor.

Och bygger stegvis upp en släktskapsmatris som i detta fall kommer bli:

Baserat på detta kan man få fram inavelskoefficienter för individerna. I exemplet ovan framgår att individer som inte är inavlade är 1 (motsvarande 100%) släkt med sig själva. När de även är inavlade så läggs det till på diagonalen. I detta fall är t.ex. individ 6 resultatet av parning mellan två halvsyskon (djur 3 och 4 som båda har djur 1 som förälder). Släktskapsgraden mellan föräldrarna 3 och 4 är då ¼ och hälften av det blir 1/8 vilket motsvarar 12,5 % inavelskoefficient för individ 6. För djur 7 är det lite mer komplext, men i tabellen framgår att släktskapet mellan dess föräldrar (5 och 6) är 7/16. Hälften av det är 7/32 och därmed blir individ 7:s inavelskoefficient knappt 22%.
Detta exempel har jag hämtat från undervisningsmaterial från en dansk forskare (Knud Christensen), och det finns med fler detaljerade steg att hitta online för den som vill dyka djupare ned i metoden;
http://www.ihh.kvl.dk/htm/kc/popgen/genetics/4/5.htm
http://www.ihh.kvl.dk/htm/kc/popgen/genetik/applets/tabularapp.htm
Referens
VanRaden, P.M. 1992. Accounting for inbreeding and crossbreeding on genetic evaluations of large populations. J. Dairy Sci. 75, 3136-3144.
https://www.sciencedirect.com/science/article/pii/S0022030292780771?via%3Dihub
Av: Susanne Eriksson, Docent och forskare vid Institutionen för husdjursgenetik SLU, 16 maj 2024